아이리스 데이터에 ctree( )를 적용하여 Species 예측 모델을 만들어보자.
> install.packages("party") > library(party) > (m <- ctree(Species ~., data=iris)) Conditional inference tree with 4 terminal nodes Response: Species Inputs: Sepal.Length, Sepal.Width, Petal.Length, Petal.Width Number of observations: 150 1) Petal.Length <= 1.9; criterion = 1, statistic = 140.264 2)* weights = 50 1) Petal.Length > 1.9 3) Petal.Width <= 1.7; criterion = 1, statistic = 67.894 4) Petal.Length <= 4.8; criterion = 0.999, statistic = 13.865 5)* weights = 46 4) Petal.Length > 4.8 6)* weights = 8 3) Petal.Width > 1.7 7)* weights = 46
만들어진 모델은 plot( )을 사용해 그림 10-5와 같이 보기 좋은 결과물로 얻을 수 있다.
> plot(m)
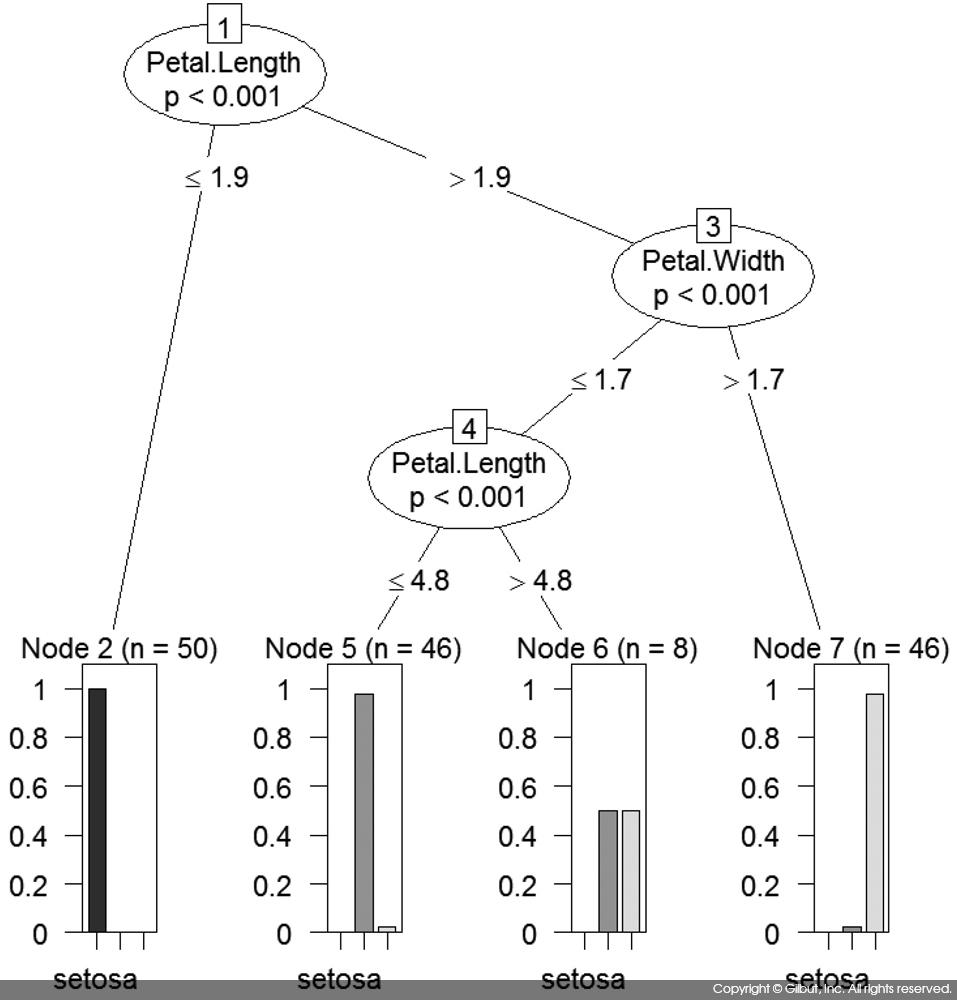
ctree( )의 결과 그림은 잎사귀 노드의 최종 분류 결과가 무엇이었는지, 또 만약 잘못 분류된 경우가 있다면 어느 정도인지를 알려주어 모델을 개선하는 데 도움을 준다. 예를 들어, 그림 10-5에서는 Node 6에 총 8개의 결과가 있는데(n=8), 이때 virginica, versicolor가 거의 동일한 숫자로 나타난 것을 알 수 있다. 따라서 이 경우를 개선하기 위한 피처나 모델을 개발한다면 성능을 더 향상시킬 수 있다.
노드 5의 경우 setosa 다음 종의 막대 그래프가 가장 높으나 그 종이 무엇인지는 그림으로 식별이 불가능하다. 두 번째 종은 iris$Species의 레벨을 확인하면 versicolor임을 알 수 있다.
> levels(iris$Species)
[1] "setosa" "versicolor" "virginica"