조건부 추론 나무
rpart를 통해 교차 검증 수행과 교차 검증 결과로부터 정확도를 계산하는 방법까지 살펴봤고, 코드가 잘 동작함을 확인했다. 다음 단계는 계속적으로 모델을 향상시키는 것으로, 크게 1) 다른 모델링 기법을 적용하거나 2) 데이터 내에 숨겨진 쓸 만한 다른 특징 값을 찾는 방법이 있다. 이 절에서는 이 중 첫 번째 방법인 다른 모델링 기법의 적용을 위해 조건부 추론 나무를 사용한다. 다음은 ctree( )를 사용한 교차 검증을 보여준다.
> library(party) > ctree_result <- foreach(f=folds) %do% { + model_ctree <- ctree( + survived ~ pclass + sex + age + sibsp + parch + fare + embarked, + data=f$train) + predicted <- predict(model_ctree, newdata=f$validation, + type="response") + return(list(actual=f$validation$survived, predicted=predicted)) + } > (ctree_accuracy <- evaluation(ctree_result)) [1] "MEAN +/- SD: 0.812 +/- 0.030" [1] 0.8119658 0.8034188 0.8135593 0.8305085 0.7881356 0.7966102 0.8050847 0.8220339 0.8813559 0.7711864
전체적인 코드의 모양은 rpart와 유사하지만, ctree는 rpart와 달리 type에 response를 지정해야 분류(dead, survived)가 반환된다는 차이가 있다. 이런 내용은 ?ctree 또는 help(library=“party”) 등의 명령을 통해 찾아볼 수 있다.
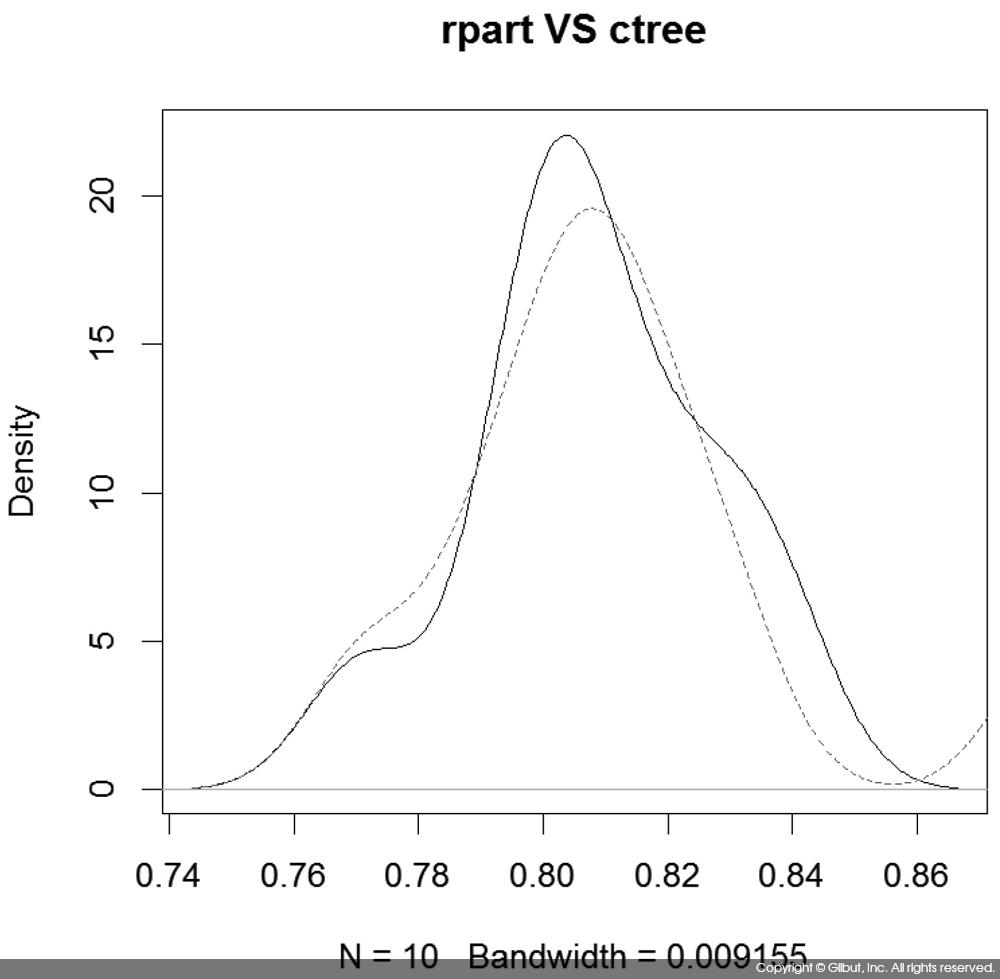
ctree( ) 수행 결과 성능이 0.812로 나타나 rpart( )의 0.808을 근소하게 앞서는 것을 볼 수 있다. 이처럼 성능이 유사한 경우 표준 편차sd 값을 눈여겨볼 필요가 있다. 만약 sd가 크다면 유의미한 성능 향상이 아닐 수도 있기 때문이다. 정확도의 분산을 비교해보면, rpart는 0.019인 반면 ctree는 0.030이었다. 표준 편차가 약 50% 정도 크기 때문에 평균 정확도는 0.004만큼 낮지만, 정확도의 값이 데이터에 따라 좀 더 크게 변동한다고 해석할 수 있다.
또는 다음과 같이 정확도 벡터에서 밀도 그림을 그려 정확도의 분포를 살펴볼 수도 있다.
> plot(density(rpart_accuracy), main="rpart VS ctree") > lines(density(ctree_accuracy), col="red", lty="dashed")